

Encuentran alarmante resistencia de bacteria en UCI de Bogotá

La genética de Pseudomona aeruginosa sería la clave para tener mejores tratamientos contra su alto impacto en la salud. Foto: Mishelle Cuello Mejía, magíster en Microbiología de la UNAL.
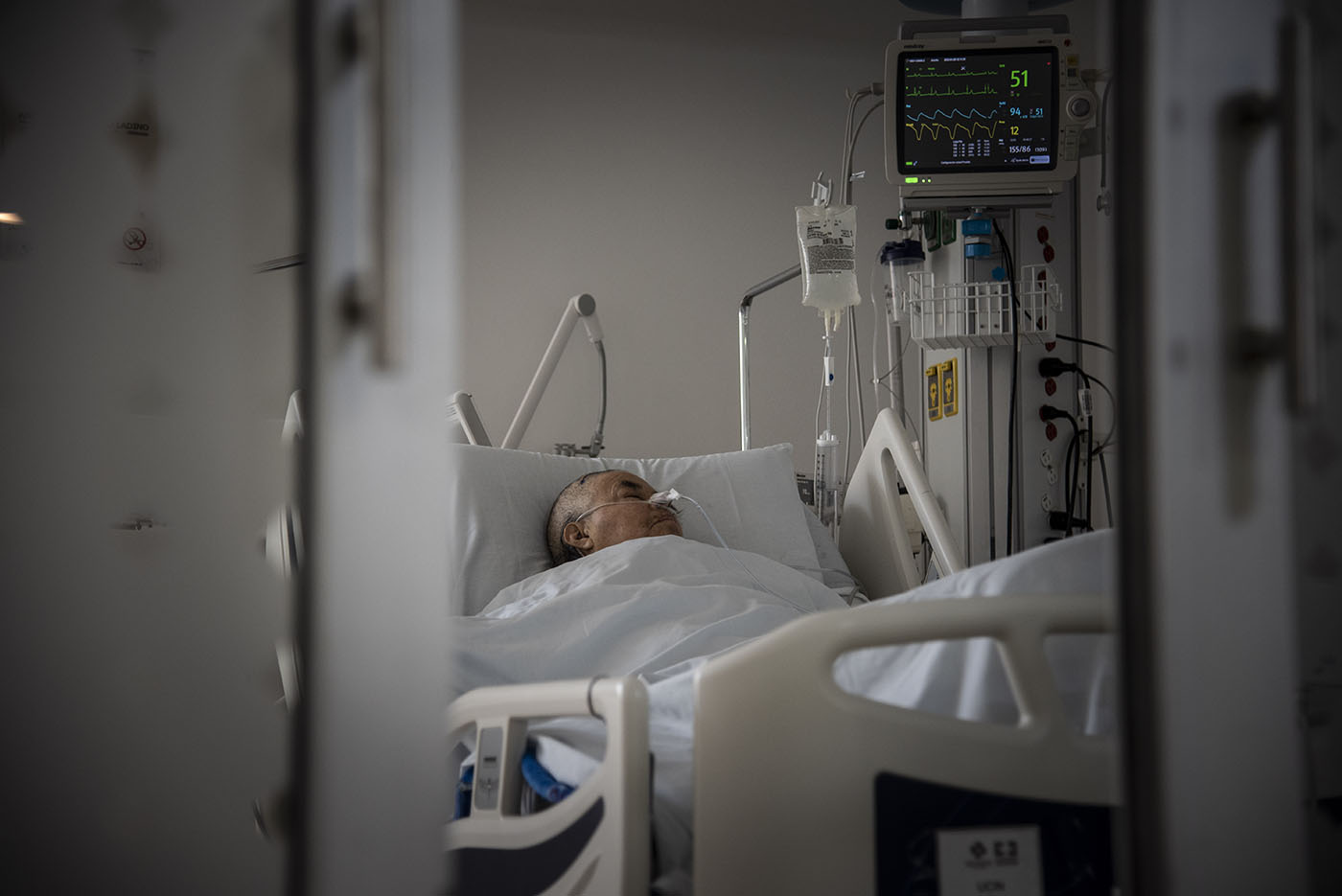
Estas bacterias presentan un gran riesgo para los pacientes en UCI, ya que se quedan a vivir allí y pueden sobrevivir largos periodos de tiempo. Foto: Jeimi Villamizar, Unimedios.

Los análisis se hicieron en secuenciadores de alto rendimiento, como el equipo Illumina MiSeq. Foto: Mishelle Cuello Mejía, magíster en Microbiología de la UNAL.
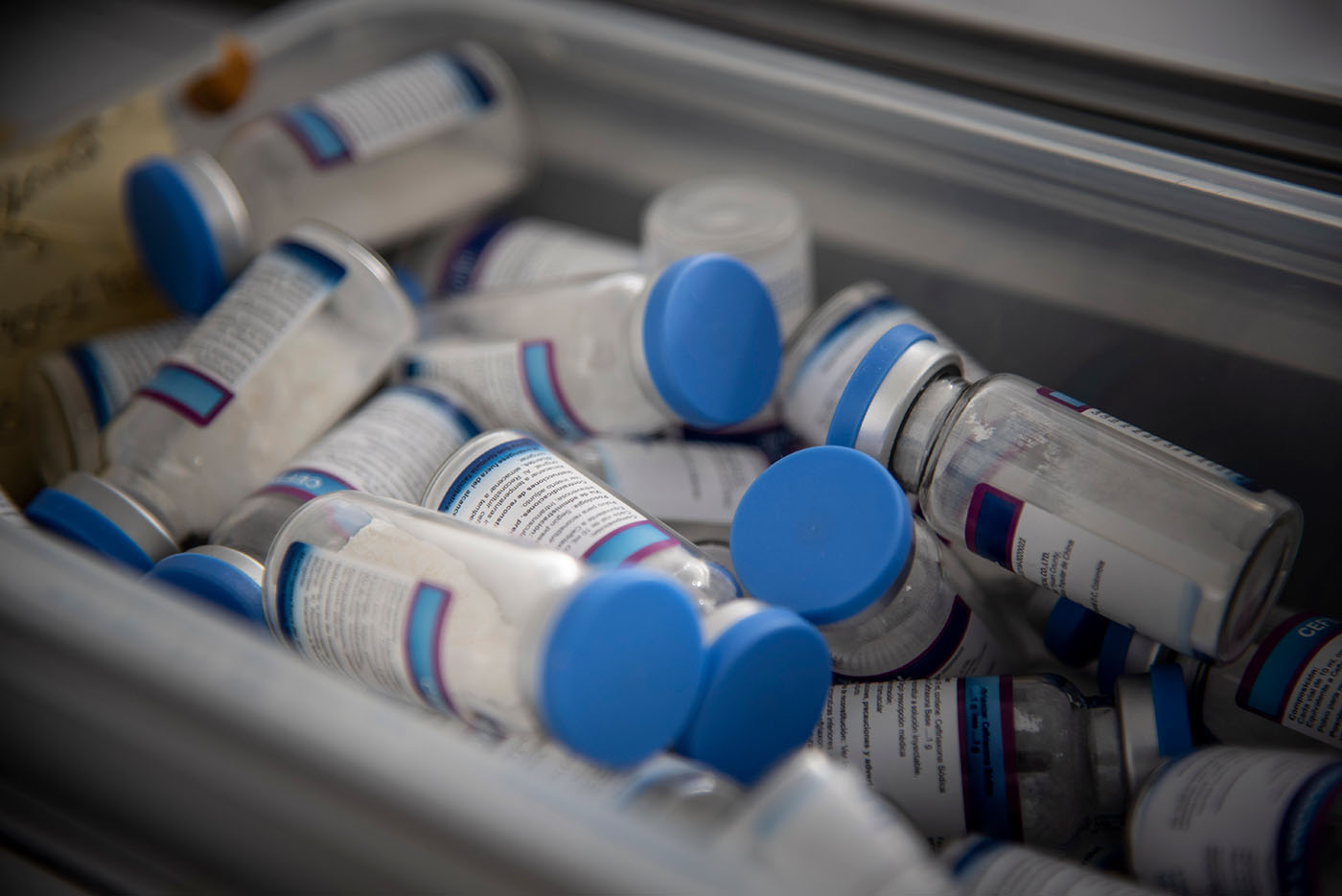
La resistencia de estas bacterias ante los antibióticos es de hasta un 67,4 %. Foto: Jeimi Villamizar, Unimedios.

Mishelle Cuello Mejía, magíster en Microbiología de la UNAL. Foto: Mishelle Cuello Mejía, magíster en Microbiología de la UNAL.
Los carbapenémicos son antibióticos utilizados como último recurso contra infecciones graves causadas por bacterias multirresistentes como P. aeruginosa, y aunque son efectivos hasta cierto punto, su acción se ve limitada por la presencia de las enzimas “carbapenemasas”, que degradan estos antibióticos.
Tal situación ha convertido las bacterias productoras de estas enzimas, en especial P. aeruginosa, en una amenaza global para la salud pública, sobre todo en las UCI.
Según la Secretaría de Salud de Bogotá, en la ciudad hay cerca de 1.096 camas de UCI habilitadas, de las cuales 846 están ocupadas en instituciones privadas y 144 en públicas. Ante esta dificultad, el estudio del genoma de estas bacterias aparece como una alternativa viable para desentrañar la forma en que se protegen y cambian, y en un futuro saber cómo atacarlas.
P. aeruginosa es un patógeno difícil de controlar en el ambiente hospitalario y puede causar problemas graves como infecciones en la sangre, la piel, los pulmones y hasta en los huesos, y su incidencia se agrava según las comorbilidades o problemas de salud de cada persona. Se ha encontrado que tienen una predilección por pacientes con quemaduras.
Tras años de estudio e investigación con estas bacterias, la microbióloga Mishelle Cuello Mejía, magíster en Microbiología de la Universidad Nacional de Colombia (UNAL), encontró dentro de un grupo de bacterias analizadas la presencia del gen blaKPC-3, que les confiere resistencia a los carbapenémicos, consolidándose como el segundo reporte en el país. Además, halló la presencia de 14 secuencio-tipos de P. aeruginosa que son nuevos en el mundo y que están circulando en Bogotá.
El estudio forma parte de un macroproyecto financiado por Colciencias –dirigido por el profesor Emiliano Barreto, de la Facultad de Ciencias de la UNAL– en el cual se hallaron tres bacterias con patrones de resistencia antimicrobiana notablemente inusuales y con un alto grado de similitud con bacterias reportadas en Shanghái, Chile y Argentina.
“Gracias a la financiación obtenida se adquirieron dos secuenciadores de alto rendimiento –de las plataformas Illumina y Oxford Nanopore– que la UNAL no tenía y que nos puso en el panorama, permitiéndonos formar parte de la Red de Laboratorios de Genómica del Instituto Nacional de Salud (INS)”, asegura la investigadora.
Para el estudio se analizaron muestras de sangre, tejidos, secreciones respiratorias y líquidos corporales de 95 pacientes en UCI, pero la tarea no fue tan fácil, pues cuando llegó el COVID-19 se restringió el ingreso a la mayoría de las instituciones hospitalarias, por lo que la microbióloga tuvo que estar muy atenta para ir por las muestras lo más rápido posible para llevarlas y analizarlas en el Laboratorio de Epidemiología Molecular de la UNAL.
“En el mismo hospital encontramos grupos de bacterias genéticamente idénticas en pacientes de camas muy distantes; por ejemplo, una bacteria identificada en un paciente de la cama 1 UCI también se halló en la cama 32 de hospitalización en otro piso, lo cual quiere decir que la bacteria viaja sin ningún problema aunque se apliquen los protocolos de desinfección e higiene”.
“Esto se relaciona con su capacidad de clonalidad y diseminación, es decir con la posibilidad de que una bacteria se divida dando origen a dos células idénticas que mantienen un mismo origen, lo cual permite determinar tanto su información genética como su procedencia. En el hospital analizado había pacientes no solo de Bogotá y Cundinamarca, sino también de Tolima, Meta y Casanare” , explica la investigadora.
El genoma de P. aeruginosa es más grande que el de la mayoría de las bacterias secuenciadas, su tamaño varía entre 5.500.000 y 7.000.000 de pares de bases, lo cual le permite tener más adaptaciones y transformaciones a lo largo del tiempo, ya que tiene una gran plasticidad genómica, mediante la cual incorpora elementos de otros microorganismos que también tienen resistencia a los antibióticos.
La secuenciación de nueva generación permite identificar sus bases y su organización, con el objetivo de determinar los genes asociados con su perfil de resistencia, todo esto de la mano de herramientas bioinformáticas que –por medio de bases de datos– organizan y catalogan esa información biológica para entender el funcionamiento y la estructura de estos microorganismos.